U nás v laboratóriu sa občas hrám s naším sekvenátorom. Je to dosť komlikovaný prístroj, ktorý stál 800 000 eur, ale ja ho už ovládam lepšie ako play-station.
Na sekvenovanie treba pripraviť DNA do určitej špeciálnej formy, aby bola nakrájaná na kúsky, ktoré sekvenátor dokáže prečítať. Tiež sa na jej koniec musí pripojiť krátka známa sekvencia DNA, aby sekvenátor vedel, kde má začať sekvenciu čítať. Táto úprava vstupnej DNA sa volá príprava sekvenačnej knižnice. Bližsie o sekvenovaní a skladaní genómov som písala už dávnejšie.
Celá príprava sekvenačnej knižnice má veľa krokov, kde sa vstupné množstvo DNA postupne stráca. Buď sa DNA prilepí na stenu skúmavky, alebo špičky pipiet, alebo sa proste nechtiac odpipetuje preč do odpadu. Tie straty ale nikomu nevadia, lebo sa začína s veľkým množstvom DNA pochádzajúcej z veľa rovnakých buniek a na samotné sekvenovanie stačí naozaj len máličko.
Výrobcovia určite vedia, že sa v celom tomto procese prípravy sekvenačnej knižnice stráca veľa DNA. Pre nich je ale dôležitejšie, aby sa celý sekvenačný systém dobre predával, a tak sa rozhodli pre jednoduché a rýchle, ale menej precízne kroky, ktoré dokáže rutinne urobiť akákoľvek zaškolená laboratórna technička. Výrobcovia jednoducho poriadne nadsadili vstupné množstvo DNA a očistili si tak ruky.
Väčšina vedcov sa, samozrejme, veľmi nezaujíma, ako vlastne prebieha príprava sekvenačnej knižnice, pretože si vzorky nechávajú sekvenovať v špecializovaných laboratóriách mimo ich pracoviska. Normálny vedec sa zvyčajne venuje biologickej stránke svojho výskumného projektu a nemôže si už dovoliť detailne študovať protokoly, ktoré sú už vopred presne nastavené výrobcom. Špecializované laboratórium povie: „Na sekvenovanie nám podľa pokynov výrobcu dodajte 1 mikrogram DNA“. Tak sa vedec ocitne v situácii, že má na jednej strane perfektne nastavený štandardný sekvenačný protokol, ktorý mu „ľavou zadnou“ urobí laboratórna technička, ale na druhej strane ma veľký problém: kde zoženiem toľko DNA?
V prípade sekvenovania baktérie z kultivačnej misky to nie je problém, pretože baktéria sa dá kultivovať vo väčšom množstve a DNA bude dosť. Robia sa však aj špeciálne vedecké projekty, kde je možné osekvenovať len zopár buniek. Napríklad v ľudskom tráviacom trakte sú milióny baktérií, z ktorých sa väčšina nedá kultivovať. Predstavte si výskumný projekt, kde sa bude sekvenovať doposiaľ nepoznaná jedna jediná bakteriálna bunka, ktorá by sa v laboratóriu nerozmnožovala. Tiež fascinujúce sú projekty typu, kde vedci skúmajú RNA z jednej jedinej rakovinovej bunky.
Môj projekt je práca s ultramaličkým množstvom DNA pochádzajúcej priamo z patogénov z tráviaceho traktu. Klasický spôsob na diagnostiku a sekvenovanie patogénov je pestovanie na Petriho miskách v špecifickom médiu. Zloženie tohoto média však môže podporovať rast nejakého bakteriálneho kmeňa, ktorý v skutočnosti v infikovaných črevách nie je až tak rozmnožený. Takže dostaneme skreslené výsledky. Dokonca možno prehliadneme nejaký novozmutovaný kmeň, ktorý v štandardných podmienkach proste nerastie vôbec. Riešením by bolo získať prírodnú populáciu tohoto patogénneho druhu, bez skreslenia kultiváciou na médiu. Vyseparovali by sme všetky bunky patriace tomuto patogénnemu druhu pomocou prietokovej citometrie, o ktorej som písala v jednom dávnejšom blogu, alebo nejakou inou separačnou technikou, ktorú práve teraz vymýšľam.
Ak majú vedci k dispozícií len veľmi malé množstvo buniek, ktoré sa nedajú namnožiť na kultivačnej miske a nevedia o našom upravenom protokole na sekvenovanie, siahnu väčšinou po špeciálnom enzýme, ktorý sa volá phi29 polymeráza. Tento enzým dokáže zvýšiť obsah DNA v skúmavke. Písala som o tom v nedávnom blogu. Je to polymeráza, ktorú pridáme do skúmavky k našej maličkej kvapke DNA a ona sa napojí na nejaký náhodný úsek DNA a začne tvoriť ďalšiu kópiu pôvodnej DNA, takže nakoniec ju veľmi veľakrát namnoží. Táto polymeráza však nepracuje na všetkých miestach genómu rovnako efektívne a okrem toho sa môže stať, že nejaké úseky preskočí alebo spojí dva úseky, ktoré k sebe nepatria. Proste čistá katastrofa! A to všetko len kvôli tomu, aby sme vyhoveli základnému požiadavku výrobcu sekvenátora, že potrebuje 1 mikrogram DNA.
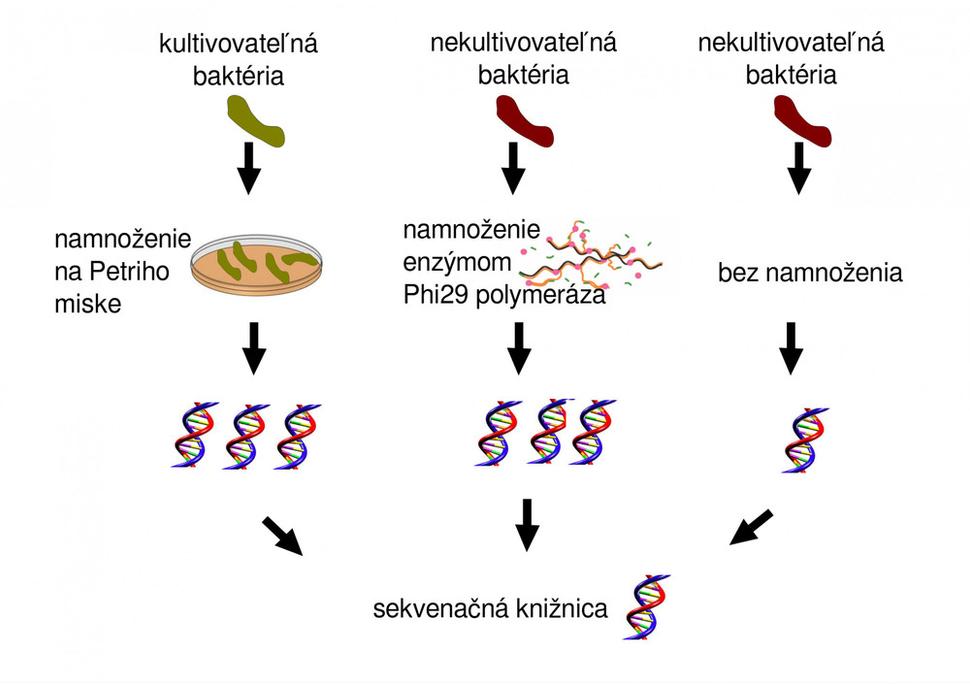
Ja pracujem s patogénmi, ktorých genómy sú veľmi variabilné a aj jednopísmenková mutácia môže mať veľký vplyv na ich nebezpečnosť, okrem toho vznikajú nové kmeňe, ktoré majú celé časti genómu totálne poprehadzované. Ja teda potrebujem naozaj pravdivé sekvencie a nemôžem dovoliť pracovať s DNA, ktorú umelo (a chybne!) namnožila phi29polymeráza.
Preto som sa rozhodla skúšať, ako by sa dali redukovať straty v jednotlivých krokoch sekvenačného protokolu. Po dlhých mesiacoch som sa dostala do bodu, že už viem „ľavou zadnou“ získať genóm baktérie, ktorý pochádza len z 5-10 tisíc baktériálnych buniek. Pre lepšie predstavenie: ak by sa teoretické množstvo DNA obsahnuté v 10000 baktériách prepočitalo na ľudské bunky, na prácu by mi stačilo len 15 ľudských buniek. Pritom na dodržanie originálneho sekvenačného protokolu daného výrobcom by som musela mať niekoľko miliónov buniek.
Takéto vylepšenia protokolu však v súčasnej dobe na poriadny vedecký článok už nestačia. Preto sme sa rozhodli obhájiť potrebu týchto vylepšení a na rovnaké množstvo baktérií E. coli aplikovali „slávnu“ phi29polymerázu. A obidve vzorky sme osekvenovali a porovnali. Mnoho kolegov, ktorí používajú phi29polymerázu mi vopred hovorili, že to občas funguje, ale občas nie. V mojom prípade to teda vôbec nefungovalo! Získali sme veľké množstvo sekvencií, ale všetko to boli absolutne nezmysly! Žiadna zhoda s genónom našej baktérie E. coli! Proste absolútne vymyslené sekvencie, ktoré na tejto planéte ešte neexistovali. Kolegovia hovorili, že je to však celkom bežné...
Kedže tieto sekvencie namnožené phi29 polymerázou nedávali žiadnu zhodu s databázami, rozhodli sme sa urobiť jednu špeciálnu bioinformatickú analýzu, aby sme zistili, odkiaľ sa tieto sekvencie asi berú. Jedna teória hovorí, že ak sa porovnajú frekvencie hexamerov v genóme, je možné zistiť nejakú podobnosť medzi druhmi. Hexamér je 6 písmen ACTG v rôznom poradí. Takže sme naše sekvencie rozkrájali na hexaméry a to sme porovnali s hexamérmi z genómov rôznych druhov baktérií. A predstavte si, že tieto nezmyselné sekvencie sa podobali na Bacillus subtilis, odkiaľ bola phi29 polymeráza vlastne prvýkrát izolovaná! Takže výrobca asi predáva nečistú phi29polymerázu, ktorá k tomu ešte ani poriadne neplní svoju funkciu namnoženia DNA.
Samozrejme, publikovalo sa už niekoľko veľmi dobrých článkov v Nature či Science, kde naozaj vedci dokázali, že v ich prípade phi29 polymeráza fungovala. Sekvencie, ktoré publikovali, sa dali dokonca poskladať do jedného celistvého genómu. Takže sa zdá, že phi29 niekedy funguje, ale niekedy nie.
Bohužiaľ, svet je zaplavený vedeckými článkami, kde sa DNA pred sekvenovaním namnožila phi29polymerázou, ale neuvádza sa žiadne overenie, či phi29 polymeráza fungovala správne. Publikujú sa čiastočné genómy, ktoré nie sú spojené do jedného celku, ale sú vo forme stoviek nespojiteľných kontigov (väčšie zhluky sekvencií).
Ja som sa nedávno podujala stiahnuť všetky genómy, ktoré boli vytvorené s použitím phi29 polymerázy a aplikovala tú istú metódu frekvencie hexamérov v týchto genómoch, aby som zistila, či sa náhodou nepodobajú na Bacillus subtilis, ktorý sa na produkciu phi29 polymerázy asi využíva. A predstavte si, že niektoré z týchto pochybných genómov mali s Bacillusom naozaj veľa spoločného! Ale to je výsledok, ktorý som prezradila vlastne len vám, ale nemôžem ho publikovať v žiadnom vedeckom časopise, lebo nechcem zhadzovať kolegov z iných laboratórií. Len poznamenám, že tie phi29 genómy z Nature a Science boli v poriadku a nemali s Bacillusom nič spoločné.
Potom som urobila ešte jednu srandovnú skúšku, ktorú tiež radšej nikde oficiálne nepublikujem. Tak, ako uvádzali v tamtých pochybných vedeckých článkoch, som moje vlastné sekvencie namnožené phi29polymerázou pospájala pomocou programu newbler. Tento bioinformatický program normálne nepoužívam, lebo je pri spájaní sekvencií veľmi benevolentný. No a predstavte si: moje nezmyselné phi29 sekvencie sa pospájali do približne 1000 kontigov! Naozaj sa v niektorých pochybných vedeckých časopisoch publikuju genómy „novoobjavených organizmov“, ktoré boli namnožené phi29 polymerázou a sú v konečnej podobe cca 1000 nespojiteľných kontigov. Ak by sa ku mne dostala na posúdenie takáto publikácia, tak ju hneď zamietnem. Keď som svoj nezmyselný genóm ukázala kolegom, tak sa začali smiať, že mám vymyslieť názov pre túto moju „novoobjavenú baktériu“. Mohla by sa napríklad volať Dzunkobacterium slovakiensis. To bol len vtip... :)
Bohužiaľ, môj boj proti použivaniu phi29 polymerázy je ťažký. Minule som na jednej konferencii stála s mojím posterom hneď vedľa posteru firmy, ktorá tento enzým predáva. Divný pocit. Aj napriek tomu, že už aj najväčšie vedecké autority na konfrenciách tvrdia, že namnoženie DNA na sekvenovanie spôsobuje viac škody než úžitku, sa firmám zjavne darí takéto enzýmy predávať. Ľudská pohodlnosť nepozná hraníc.
Nezmyselné genómy stvorené phi29 polymerázou sa našťastie publikujú „len“ vo vedeckých časopisoch, ale nenechádzajú sa, chvalabohu, v oficiálnych databázach sekvencií ako referenčné genómy. Je škoda, že mnohí kolegovia iba bezhlavo plnia požiadavky výrobcov sekvenátorov a nezmyselne si namnožia svoje DNA vzorky týmto nespoľahlivým enzýmom. Pritom je tak jednoduché naučiť sa používať náš upravený protokol na sekvenovanie ultramaličkého množstva DNA...






